| 技術名稱 |
傾聽來自生物大數據的浪潮 (SeaShell):單細胞RNA定序實驗資料之自動化分析平台 |
| 計畫單位 |
中央研究院 |
| 計畫主持人 |
林仲彥 |
| 技術簡介 |
近年來,隨著單細胞分選技術以及基因定序偵測極限的突破,單細胞RNA定序(Single-cell RNA sequencing, scRNA-seq)技術讓科學家能夠自由探索每顆細胞獨有的基因表現特徵,將轉錄體學的研究推進至傳統定序實驗無法比擬的高解析度分析層次。因此不意外地,這門技術被廣泛地應用於全世界最前端的生物醫學領域研究,如再生醫學、轉譯醫學與精準醫療等。
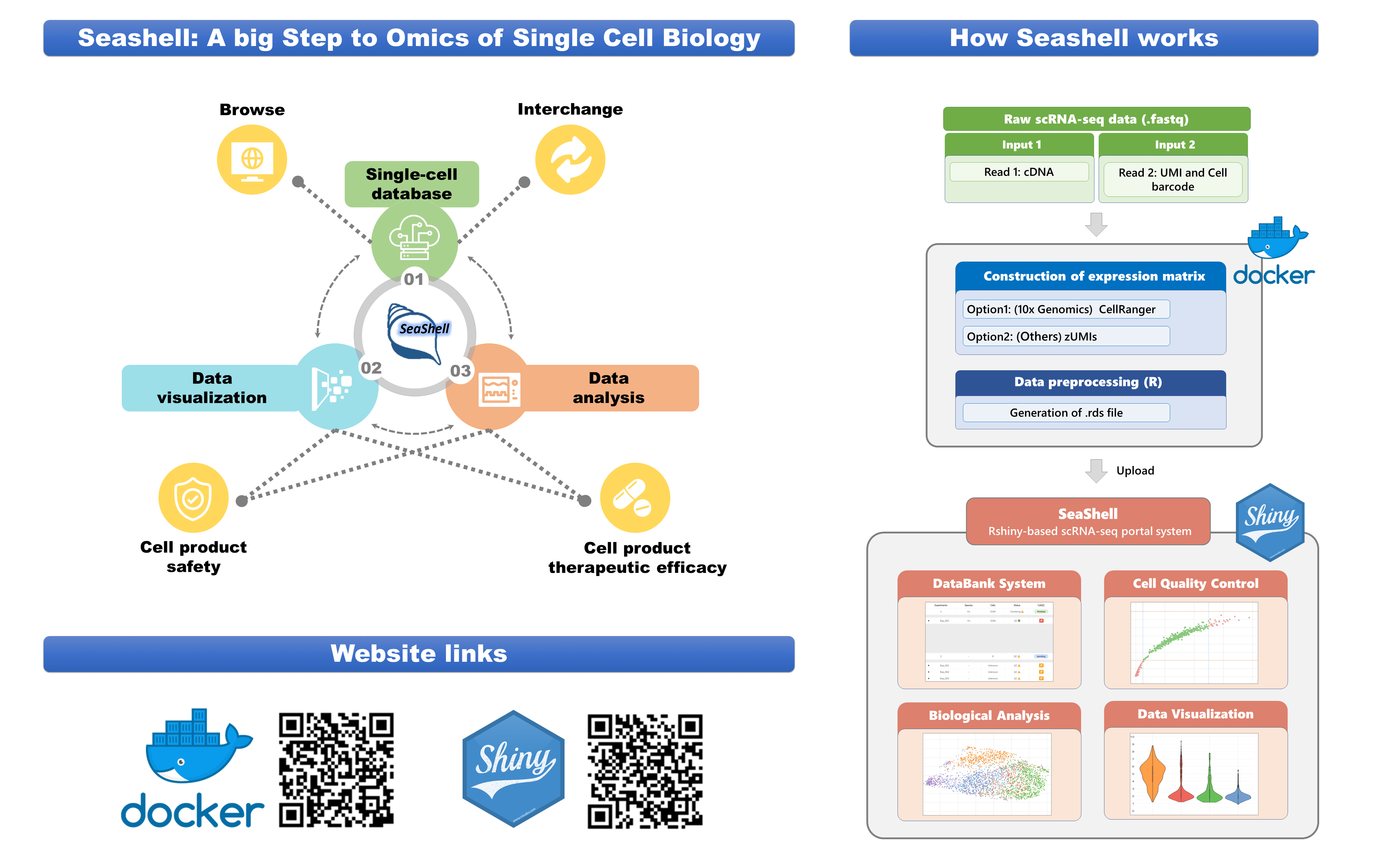
然而,高解析度所帶來的高維度單細胞基因定序資料卻也提高了後續分析的難易度,使得資料分析成為進行單細胞定序實驗的一大技術門檻。因此,本研究團隊希望藉由提供一自動化的單細胞RNA定序實驗分析平台,SeaShell,來協助生物醫學領域的學者從龐大的基因資料中取得有意義的資訊。SeaShell分析平台可分為兩個部分:
(一)Docker-Galaxy資料前處理平台
本研究團隊應用Docker虛擬化技術以及Galaxy生物資訊開源平台,將單細胞定序原始資料(fastq檔案)的前處理流程簡化為直覺化的圖形化操作介面。使用者可以隨時根據自身的研究需求與實驗設計,自行進行以往不容易處理的原始資料之前處理與參數調整,並取得單細胞基因量表現資料以及能夠於後續網頁平台分析之R語言資料檔案(rds檔案)。
(二)RShiny自動化之網頁分析與視覺化平台
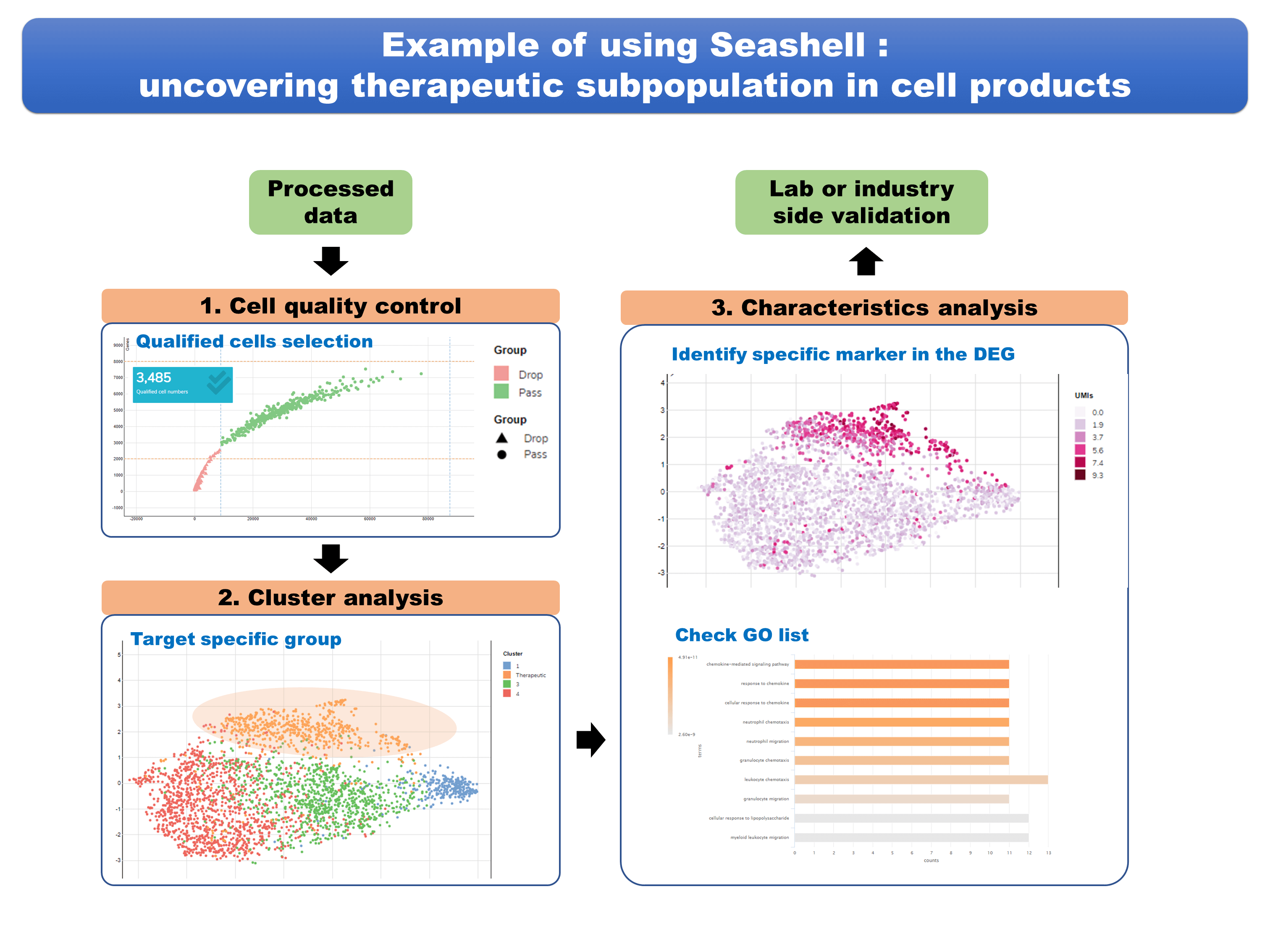
本研究團隊以單細胞分析工具中最常使用的R語言為基礎,搭配RShiny、Java Script D3等網頁視覺化套件設計此網頁平台,其主架構可分為三大部分:資料庫(Databank)、資料分析(Data analysis),以及資料視覺化(Data visualization)。使用者可透過直觀操作的圖形化介面,自由地搭配各種單細胞RNA定序分析工具來進行分析。例如:針對同一分析計畫中的所有子定序實驗資料進行細胞品質管控、正規化、群集分析、差異性表現基因分析、生物標靶基因分析、GO分析等標準化資料分析流程。除此之外,此平台的一大特色在於使用者亦可客製化地調整流程中之分析參數,取得最佳的資料分析結果後再儲存於資料庫中,並透過多樣化的資料視覺化平台隨時讀取前次之最佳分析結果。
簡言之,SeaShell自動化分析平台可顯著降低單細胞RNA表現之分析技術門檻,讓研究者可以透過大數據的協助,快速瞭解大量細胞組成與功能調控,來推進最新生物醫學研究以及相關產業發展。 |
| 科學突破性 |
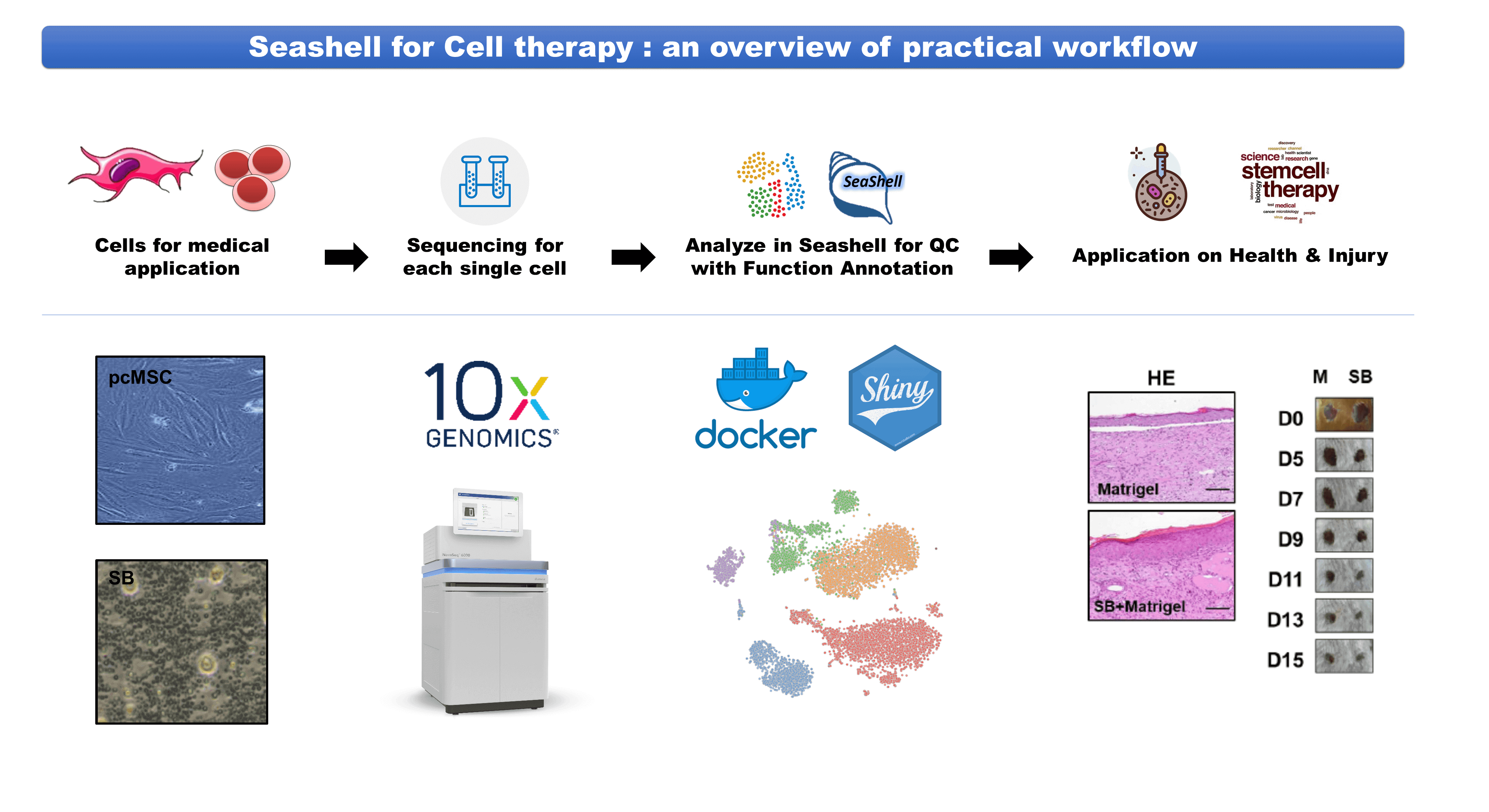
此一單細胞RNA定序資料平台的特色主要可列出三點:(一)從原始資料開始的客製化分析流程(二)直覺化的資料庫介面(三)多樣化的整合性視覺化平台。本團隊亦與臺北醫學大學以及臺灣大學醫學院之團隊合作,將此平台應用於細胞治療產品之有效細胞群體分析,成功找出可能與療效有關的細胞群體,證明了此分析平台的實用性。 |
| 產業應用性 |
在產業應用方面,本研究團隊正與臺北醫學大學、臺灣大學醫學院的臨床細胞醫療團隊進行密切合作,以單細胞RNA定序實驗取得每一顆細胞之高維度基因資訊,並以本團隊開發的網頁平台Seashell進行後續分析,為這些細胞產品提供了相當於「細胞藥物仿單」的安全性資訊。未來將推廣此平台至更多不同研究領域及產業應用。 |
| 關鍵字 |
單細胞定序 轉錄體分析 基因定序 自動化分析 細胞銀行 細胞治療 細胞藥物仿單 生物標靶分析 精準醫療 基因大數據 |
| 備註 |
分析程式平台與網站連結:
Docker-Galaxy資料前處理平台:
https://hub.docker.com/r/lsbnb/galaxy_sc_20200421
自動化之網頁分析與視覺化平台:
http://seashell.iis.sinica.edu.tw/seashell/ |